Precise microbiome engineering using natural and synthetic bacteriophages targeting an artificial bacterial consortium
https://www.frontiersin.org/journals/microbiology/articles/10.3389/fmicb.2024.1403903/full
【研究概要】
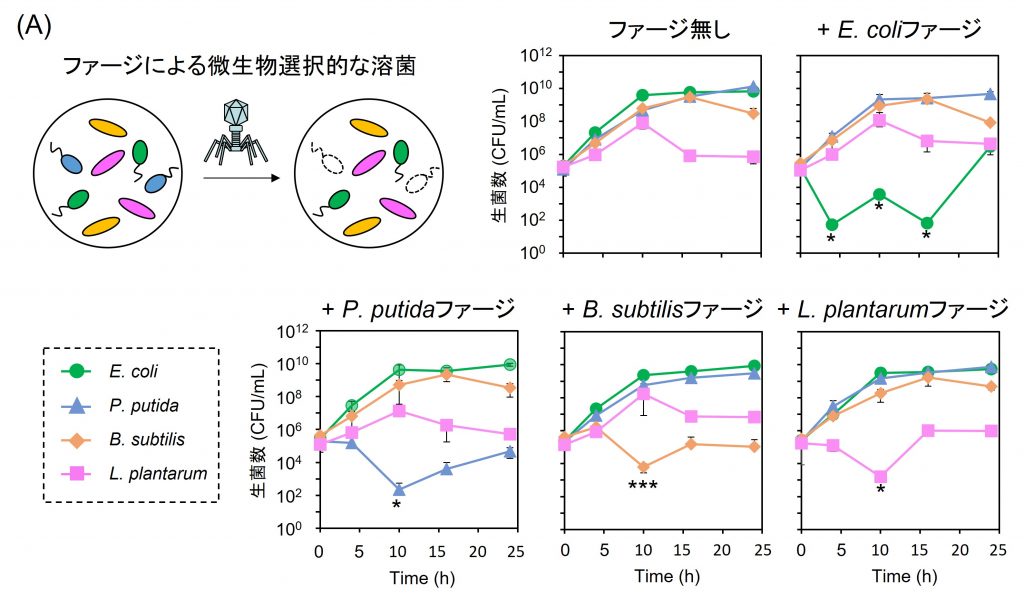
微生物の集合体である菌叢は、宿主の健康改善や土壌の元素循環などに重要です。菌叢の働きを自在に改変・制御できれば、健康寿命の延伸や持続的耕地利用といった社会課題の解決に貢献できると期待できます。そこで、本論文ではバクテリオファージが有する宿主特異的な溶菌能力に着目し、菌叢においてファージを用いた菌種特異的な増殖抑制が可能であるかを評価しました(図A)。
菌叢としては、実菌叢での検証に先駆けて、Eshcerichia coli、Pseudomonas putida、Pseudomonas fluorescens、Lactiplantibacillus plantarumの4菌からなる人工菌叢を用いました。この人工菌叢に各微生物に感染するファージを添加した結果、標的とする微生物の菌数だけを減らすことに成功しました(図A)。しかしながら、いずれの微生物においてもいったん菌数が減少した後、菌数の上昇が確認されました。これは、ファージ耐性菌の発生によるものであり、今後の課題となります。
上記の実験は自然界から単離したファージを用いて行いましたが、ファージの単離には大変な手間を要します。ファージの中には溶原ファージという自身のゲノムを宿主のゲノムに潜伏させる能力を有するものがいます。この潜伏したファージDNA(プロファージと呼ぶ)はファージの設計図に他ならず、プロファージからファージを人工合成できないか?というアイデアに至りました。そこで、本コンセプトを実証すべくλファージが溶原化したE. coliのゲノムを鋳型として溶原化に関連する遺伝子を除くファージDNA断片をPCR増幅しました。この際、各DNA断片間が50 bp程度のオーバーラップ配列を持つようにPCR増幅し、これをE. coliにヒートショック法で導入しました。E. coliに相同組換え酵素を発現しておくことでファージDNAの断片をつなぎ合わせることができ、全長のファージDNAをin vivoで合成することができます。そして、E. coliがファージDNAを転写・翻訳することで、ファージの単離を経ることなくファージを人工合成することができます。合成したファージを用いて菌叢改変実験を行った結果、天然ファージと同様、菌叢中のE. coliの菌数を減らすことに成功しました。
今後は、実菌叢中の微生物を標的とするファージを探索あるいは人工合成することで、実菌叢の改変を行い、菌叢微生物の機能解明や機能制御を行っていきたいと考えています。

